El ADN puede señalar la presencia o la predisposición a una gran cantidad de enfermedades, incluido el cáncer. La capacidad de detectar estas pistas, conocidas como biomarcadores, permite a los profesionales médicos realizar diagnósticos tempranos críticos y brindar tratamientos personalizados. Los métodos típicos de detección pueden ser laboriosos, costosos o limitados en lo que pueden descubrir. Un nuevo chip biosensor que cuenta con un diseño preciso y económico puede aumentar la accesibilidad a diagnósticos de alta calidad.
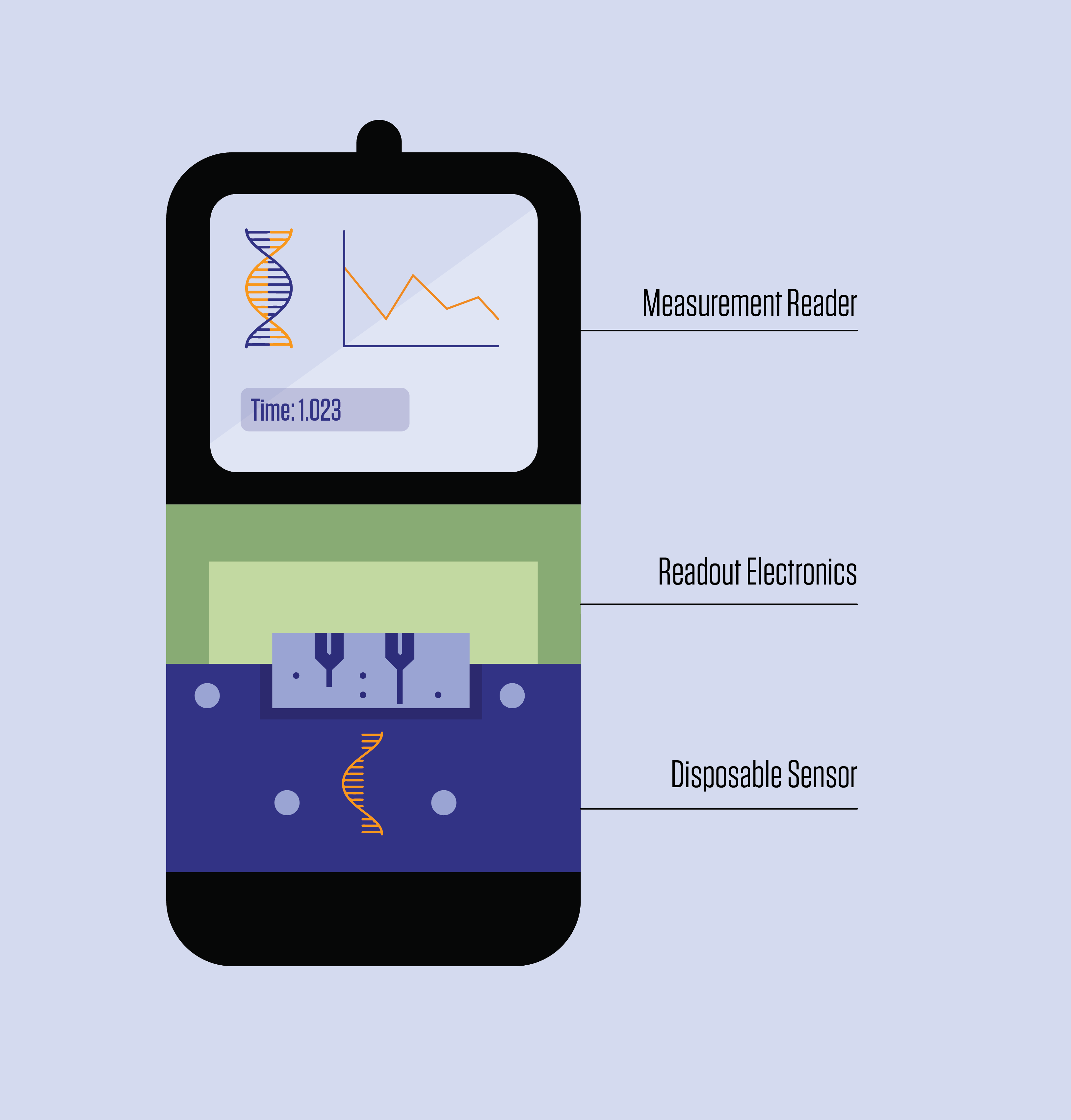
El biosensor, desarrollado por investigadores del Instituto Nacional de Estándares y Tecnología (NIST), la Universidad de Brown y el instituto de investigación financiado por el gobierno francés CEA-Leti, identifica biomarcadores midiendo cómo se unen las hebras de ADN y el dispositivo. Lo que lo diferencia de otros sensores similares es su diseño modular, que reduce los costos al facilitar la producción en masa y permitir la reutilización de los componentes más costosos.
En un documento recién publicado en línea de la última Reunión Internacional de Dispositivos de Electrones de IEEE, los investigadores presentaron los resultados de un estudio que demuestra la alta sensibilidad y precisión del dispositivo a pesar de su modularidad, que generalmente se asocia con un rendimiento reducido.
Al igual que otros biosensores de ADN, el dispositivo aprovecha el hecho de que una sola hebra de ADN, cuando no está emparejada con otra dentro de la doble hélice familiar, está preparada para la unión química. Parte del dispositivo está recubierta con hebras simples de ADN. Cuando estas «sondas» encuentran biomarcadores de ADN que tienen una secuencia genética correspondiente o complementaria, las dos hebras se unen y envían una señal que es captada por el dispositivo.
“Para hacer la medición, necesitamos dos moléculas de ADN. Colocamos una hebra en nuestro sensor que es complementaria al ADN objetivo, esa es la proverbial aguja en el pajar”, dijo el investigador del NIST Arvind Balijepalli, coautor del nuevo estudio.
Cuando una cadena de ADN objetivo se une a una sonda, induce un cambio de voltaje que un dispositivo semiconductor, llamado transistor de efecto de campo (FET), puede medir. Estos cambios de voltaje pueden ocurrir cientos de veces por segundo a medida que las moléculas entran y salen del sensor.
Debido a su alta resolución de tiempo, este enfoque puede decirle no solo si una hebra de ADN está unida a una sonda, sino también cuánto tarda en conectarse y desconectarse, un factor llamado cinética de unión que es clave para discernir diferentes marcadores que pueden unirse a la misma sonda en diversos grados.
Y con este método, no necesita mucho espacio para medir mucho.
“Esta es una técnica muy escalable. En principio, podemos tener cientos, si no miles, de sensores en un área de un milímetro cuadrado integrados en un dispositivo del tamaño de un teléfono inteligente, que es mucho menos engorroso que algunas de las tecnologías que se usan actualmente en la clínica”, dijo Balijepalli.
Sin embargo, los métodos basados en FET aún no se han generalizado. Un escollo importante es su carácter de un solo uso, que hasta ahora parecía una necesidad pero incrementa su coste.
De manera similar a cómo su radio se vuelve cada vez más ruidosa a medida que se aleja de una estación de radio, las señales eléctricas también se vuelven más ruidosas cuanto más tiempo tienen que viajar dentro de la electrónica. El ruido aleatorio no deseado recogido en el camino hace que la señal sea más difícil de medir.
Para limitar el ruido, las sondas de ADN en los sensores basados en FET normalmente se conectan directamente al transistor, lo que convierte la señal en datos legibles. El inconveniente es que las sondas se gastan después de exponerse a una muestra y, por lo tanto, todo el dispositivo también.
En el nuevo estudio, Balijepalli y sus colegas aumentaron la distancia entre las sondas y el transistor para poder reutilizar los elementos más costosos del circuito. La penalización inicial fue que la distancia podría aumentar la cantidad de ruido; sin embargo, había mucho que ganar con la elección del diseño, incluso más allá del ahorro de costos.
“Si el lector es reutilizable, podemos incorporar una tecnología más sofisticada y obtener una mayor precisión de las lecturas, y puede interactuar con el elemento de detección económico y desechable”, dijo Balijepalli.
Debido a que anticiparon que el diseño modular disminuiría la sensibilidad del biosensor, los investigadores tomaron una página del libro de jugadas de Internet de las cosas (IoT), que se adapta a las pérdidas asociadas con los dispositivos inalámbricos. Los autores del NIST combinaron su circuito con un tipo específico de FET de potencia extremadamente baja desarrollado en CEA-LETI que se utiliza en relojes inteligentes, asistentes personales y otros dispositivos para amplificar señales y compensar la sensibilidad perdida.
Para probar el rendimiento de su dispositivo, lo colocaron en muestras líquidas que contenían hebras de ADN asociadas con la exposición a radiaciones ionizantes nocivas. Sondas de ADN complementarias adornadas con electrodos conectados al FET. En varias muestras, variaron la cantidad de ADN objetivo.
Los investigadores descubrieron que la cinética de unión era lo suficientemente sensible como para realizar mediciones precisas incluso en concentraciones bajas. En general, el rendimiento del diseño modular coincidió con el de los biosensores integrados basados en FET no modulares.
El próximo paso en su investigación es averiguar si su sensor puede funcionar de manera similar con diferentes secuencias de ADN causadas por mutaciones. Dado que muchas enfermedades están causadas o asociadas con el ADN mutado, esta capacidad es esencial para el diagnóstico clínico.
Otros estudios pueden evaluar la capacidad del sensor para detectar material genético asociado con virus, como el COVID-19, que podría insinuar una infección.
Mientras tanto, la nueva tecnología podría representar una base viable sobre la cual construir.
“Existe la oportunidad de desarrollar sensores modulares más sofisticados que sean mucho más accesibles sin sacrificar las mediciones de alta calidad”, dijo Balijepalli.
Papel: Seulki Cho, Alexander Zaslavsky, Curt A. Richter, Jacob M. Majikes, J. Alexander Liddle, François Andrieu, Sylvain Barraud y Arvind Balijepalli. Mediciones de cinética de unión de ADN de alta resolución con transistores FD-SOI de doble puerta. Reunión Internacional de Dispositivos Electrónicos 2022. Publicado en línea el 23 de enero de 2023. DOI: 10.1109/IEDM45625.2022.10019493
